Μια νέα μελέτη, παρουσίασε πρόσφατα το UNIST (Ulsan National Institute of Science and Technology), η οποία αφορά σε ένα νέο στατιστικό αλγόριθμο, ικανό να εντοπίσει πιθανά γονίδια ασθενειών με πιο ακριβή και οικονομικά αποδοτικό τρόπο. Αυτός ο αλγόριθμος θεωρήθηκε ως μια νέα υποσχόμενη προσέγγιση για την ταυτοποίηση γονιδίων υποψήφιων ασθενειών, καθώς λειτουργεί αποτελεσματικά με λιγότερα γονιδιωματικά δεδομένα και διαρκεί μόνο ένα ή δύο λεπτά για να δώσει αποτελέσματα.
Την ανακάλυψη έκανε ο καθηγητής Dougu Nam και την ερευνητική ομάδα του στη Σχολή Επιστήμη της Ζωής στο UNIST. Τα ευρήματά τους δημοσιεύθηκαν στο επιστημονικό έντυπο «Nucleic Acids Research».
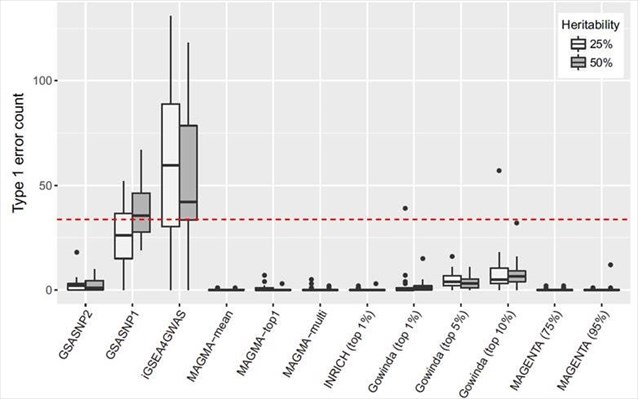
Στη μελέτη, η ερευνητική ομάδα παρουσίασε τη νέα μέθοδο και το λογισμικό GSA-SNP2 για τον εμπλουτισμό της ανάλυση οδού των δεδομένων GWAS P (Genome-wide association studies). Σύμφωνα με την ερευνητική ομάδα, το GSA-SNP2 παρέχει πολύ υψηλά αποτελέσματα στον έλεγχο σφάλματος τύπου Ι και γρήγορο υπολογισμό με την ενσωμάτωση του τυχαίου μοντέλου και του αριθμού των γονιδίων που έχουν ρυθμιστεί με το SNP.
«Το GSA-SNP2 είναι ένα ισχυρό και αποτελεσματικό εργαλείο για τον εμπλουτισμό της γνώσης για την πορεία των γονιδιων και την ανάλυση του δικτύου των γενικών δεδομένων για τη μελέτη συσχετισμού γονιδιώματος (GWAS)» λέει ο καθηγητής Nam και συμπληρώνει. «Με αυτόν τον αλγόριθμο, μπορούμε να κατανοήσουμε πιο εύκολα την ασθένεια και να την ξεκλειδώσουμε με στόχο να φτιάξουμε νέες θεραπείες για να την αντιμετωπίσουμε».
UNIST
Σύμφωνα με τον καθηγητή, το γονιδίωμα κάθε ατόμου είναι ένας μοναδικός συνδυασμός αλληλουχιών DNA που παίζουν μείζονα ρόλο στον προσδιορισμό του ποιοι είμαστε. Αυτό αντιπροσωπεύει όλες τις μεμονωμένες διαφορές, συμπεριλαμβανομένης της επιδεκτικότητας του κάθε ανθρώπου για ασθένεια και την ποικίλων φαινοτύπων. Τέτοιες γενετικές παραλλαγές μεταξύ των ανθρώπων είναι γνωστές ως πολυμορφισμοί απλού νουκλεοτιδίου (SNPs) - Single nucleotide polymorphisms SNPs (pronounced “snips”). Οι πολυμορφισμοί ενός νουκλεοτιδίου, που συχνά ονομάζονται SNPs (προφέρονται "snips"), είναι ο πιο συνηθισμένος τύπος γενετικής ποικιλότητας μεταξύ των ανθρώπων.
Τα SNP που σχετίζονται με συγκεκριμένες ασθένειες θα μπορούσαν να χρησιμεύσουν ως προγνωστικοί βιοδείκτες για να βοηθήσουν στην ανάπτυξη νέων φαρμάκων. Μέσω της στατιστικής ανάλυσης των συνοπτικών δεδομένων GWAS, είναι δυνατόν να εντοπιστούν τα νουκλεοτίδια που σχετίζονται με τη νόσο.
UNIST
Παρά τα αστρονομικά ποσά και τον απίστευτο χρόνο που επενδύθηκαν στη στατιστική ανάλυση των δεδομένων των SNP, οι συμβατικές τεχνολογίες ανίχνευσης δεν μπόρεσαν να εντοπίσουν όλους τους δείκτες των νουκλεοτιδίων. Αυτό οφείλεται στο γεγονός ότι οι περισσότερες από τις συμβατικές μεθόδους για την ανίχνευση SNPs είναι σχεδιασμένα για να ελέγχουν αυστηρά τα ψευδώς θετικά στα αποτελέσματα. Ως εκ τούτου, μεταξύ δεκάδων χιλιάδων δεδομένων γονιδιωματικής και εκατοντάδων χιλιάδων SNP που αναλύθηκαν, ο αριθμός των δεικτών που περιγράφονται σε ένα υποψήφιο γονίδιο νόσου συχνά φθάνει σε μερικές δεκάδες.
«Αν και ο έλεγχος των ψευδώς θετικών SNP είναι απαραίτητος για τη σωστή ερμηνεία των αποτελεσμάτων, το υπερβολικό φιλτράρισμα μπορεί να παρεμποδίσει τη χρησιμότητά τους στην ανάπτυξη φαρμάκων», λέει ο καθηγητής Nam. «Για το λόγο αυτό, η ενισχυμένη στατιστική ισχύς είναι απαραίτητη για τους πρακτικούς στατιστικούς αλγόριθμους».
Η ομάδα στόχευε στην ανάπτυξη ενός αλγορίθμου που θα βελτιώνει την στατιστική προβλεψιμότητα διατηρώντας ταυτόχρονα τον ακριβή έλεγχο των ψευδώς θετικών αποτελεσμάτων. Για να γίνει αυτό, εφάρμοσαν την μονοτονική καμπύλη τάσεων Cubic Spline στο γονιδιακό σκορ μέσω της ανάλυσης ανταγωνιστικής οδού για δεδομένα γονιδιακής έκφρασης.
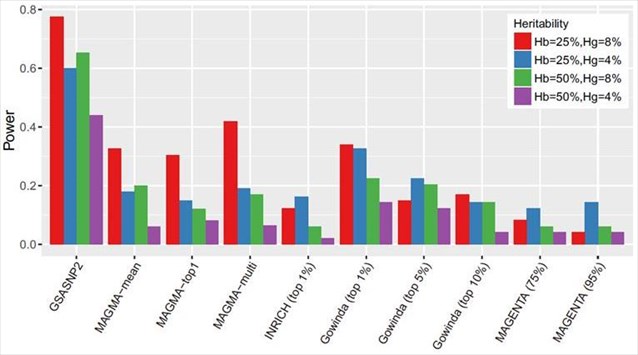
Σε μια συγκριτική μελέτη προσομοίωσης με πραγματικά δεδομένα GWAS, η GSA-SNP2 παρουσίασε θετικά αποτελέσματα υψηλής ισχύος και βέλτιστης προτεραιότητας σε σχέση με έξι υπάρχουσες μεθόδους εμπλουτισμού και δύο αυτοτελείς μεθόδους. Με βάση αυτά τα αποτελέσματα, ερευνήθηκε η διαφορά μεταξύ των προσεγγίσεων ανάλυσης του “μονοπατιού” και συζητήθηκαν οι επιδράσεις των δομών του γονιδίου συσχέτισης για περαιτέρω διερεύνηση. Επιπλέον, το GSA-SNP2 είναι σε θέση να απεικονίσει δίκτυα αλληλεπίδρασης πρωτεΐνης εντός και μεταξύ των σημαντικών οδών έτσι ώστε ο χρήστης, να μπορεί να δώσει προτεραιότητα τα βασικά υποδίκτυα για περαιτέρω μελέτες.
Σύμφωνα με την ερευνητική ομάδα, το GSA-SNP2 παρέχει έναν πολύ βελτιωμένο έλεγχο σφάλματος τύπου Ι χρησιμοποιώντας τα αποτελέσματα των γονιδίων με ρύθμιση SNP, διατηρώντας παράλληλα υψηλή στατιστική ισχύ. Επίσης, όπως ανέφεραν οι ερευνητές, το GSA-SNP2 είναι σε θέση να απεικονίσει όλα δίκτυα αλληλεπίδρασης πρωτεϊνών εντός και πέρα από τις σημαντικές οδούς έτσι ώστε ο χρήστης να μπορεί να δώσει προτεραιότητα στα βασικά υποδικτυακά δίκτυα για περαιτέρω μελέτες.